構造ファイルの新規保存
分子モデルの修正の過程で、各分子(蛋白質鎖、水分子、リガンド分子など)に対応した複数のmsfファイルが開かれていることがある(図5.1.1)。
CNX Interfaceの実施の前に、これらを一つのmsfファイルにまとめる。
|
|
(図5.1.1) “Molecule Manegement” 蛋白質鎖(kn_B1_refine)、X-Solventで挿入した水分子(searchwaters)、リガンド(heme)が、異なるmsfファイルとして扱われている状態。 |
-
“QUANTA”メニュー“File”→“Save as”を選択。
⇒“Choose the writing options”が表示(図5.1.2)。
-
“OK”をクリック。
*チェック項目はdefault設定で可。
⇒“Save Select Atoms to MSF”が表示(図5.1.3)。
-
任意の名称(拡張子:msf、例では“kn_B1S1.msf”)を入力し、“Save”。
⇒ファイル名が更新(図5.1.4)。
*複数のモデル(msfファイル)が一つのファイルに新規保存されただけで、“QUANTA”画面上の構造に変化はない。
 (図5.1.2) “Choose the writing options” |
 (図5.1.3) “Save Select Atoms to MSF” |
(図5.1.4) 分子モデルの新規保存 全分子が1ファイルにまとめられる。 |
構造ファイルの編集
分子モデルの修正過程で起こるファイル形式の乱れの修正、新規挿入した分子情報の入力、などを行う。
なお、モデルに含まれる分子によって、作業手順が異なる。
分子モデルが蛋白質のみ場合は、2. 6.の作業は不要。
-
“QUANTA”メニュー“Edit”→“Split/Clean”を選択。
⇒“Clean MSFs”パレットが表示され(図5.1.5)、分子モデルの表示が変わる(図5.1.6)。
-
“Clearn MSFs”パレット“Save to Separate MSFs”をクリック。
⇒新たに各分子毎の構造ファイルへと分割される(図5.1.7)。
-
“Clean MSFs”パレット“Clean Options”をクリック。
⇒“Protein Clean Option”が表示(図5.1.8)。
-
“Add Hydrogens”→“None”を選択し“OK”.
-
“Clearn MSFs”パレット“Clean Protein”をクリック。
-
“Clearn MSFs”パレット“Clean Solvent”をクリック。
-
“Finish”で“Split/Clearn”を終了。
⇒画面上の分子表示が元の状態に戻る。
|
(図5.1.5) “Clean MSFs” |
(図5.1.6) “Split/Clean”の実行 各分子が色違いで表示される。 (緑: 蛋白質分子、青:水分子、赤:その他) |
|
リガンドを含む場合
分子モデルにリガンドなどの分子が含まれる場合、上記に加え“Molecular Editor”での作業が必要となる。
|
(図5.1.9) 構造ファイルの選択 |
|
(図5.1.10)“Molecular Editor” |
|
-
“Molecular Editor”パレット“Edit Atoms”をクリック。
⇒“Edit Atoms”パレットが表示(図5.1.13)。
-
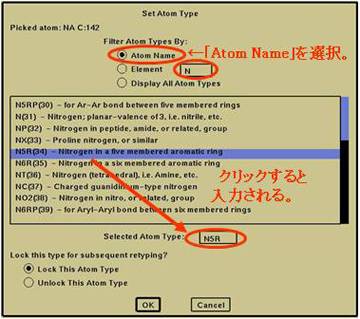
“Set Atom Type”をクリックし、“QUANTA”画面上のリガンドを構成する原子をクリック。
⇒“Set Atom Type”が表示(図5.1.14)。
-
“Set Atom Type”の“Atom Name”に適切な原子名を、また“Selected Atom Type:”に適切な“Atom Type”を入力し、“OK”。
⇒ “Atom Type”が変更される。
-
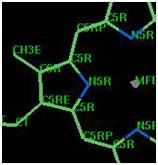
5.〜7.の操作を全原子で実行する(図5.1.15)(参考:“Atom Type”の入力)。
 (図5.1.13) “Edit Atoms” |
 (図5.1.14) “Set Atom Type” |
 (図5.1.15) “Set Atom Type”の終了 |
-
“Edit Atoms”パレット“Exit Edit Atoms”をクリック。
⇒“Edit Atoms”パレットが閉じる。
-
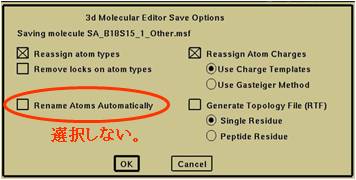
“Molecular Editor”パレット“Save and Exit”をクリック。
⇒“3d Molecular Editor Save Options”が表示(図5.1.16)。
-
設定を確認の上、“OK”。
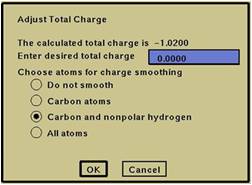
⇒“Adjust Total Charge”が表示(図5.1.17)。
-
設定を確認の上、“OK”。
⇒“Molecular Editor”パレットが閉じる。
*“Enter desired total charge”の入力値は、“The calculated charge is”での計算値(例では-1.0200)で可(?)。
未設定の原子がある場合、図5.1.18が表示(参考:“Atom Type”の入力)。
-
“Split/Clearn”を終了(“Finish”)。
 (図5.1.16) “3d Molecular Editor Save Options” |
 (図5.1.17) “Adjust Total Charge” |
以上で、CNX Interfaceを実行する準備が終了。
参考: “Atom Type”の入力
リガンドを構成する各原子には、適切な“Atom Type”を設定する必要がある。
これらが正しく設定されていないと、CNX Interfaceを実行後リガンドの構造が本来の構造から大きく逸脱してしまう。
“Atom Type”は各リガンドの構造を確認し、一覧から適当と思われるものを選択する。
例えば、図5.1.12(c)の窒素原子は、defaultでは“NT(四面体を構成する窒素)”とされているが、
新たに“N5R (五員環を構成する窒素)”を選択、設定し直している(図5.1.15)。
なお、“Atom Type”が設定されていない原子がある場合、図5.1.18の様なメッセージが表示される。
“Abort”をクリックすると未設定の原子が確認(図5.1.19)できるので、適切に設定する。
 (図5.1.18) 未設定原子のメッセージ |
 (図5.1.19) 未設定原子の表示 |


